Un microchip stana le resistenze agli antibiotici
Ricercatori bernesi hanno messo a punto una pulce elettronica che permette di individuare rapidamente dei batteri resistenti agli antibiotici.
Il professore Giorgio Zanetti, epidemiologo all’ospedale universitario di Losanna, è persuaso che questa è la tecnologia del futuro.
I ricercatori dell’Istituto di batteriologia veterinaria dell’Università di Berna hanno impiegato tre anni per sviluppare un microchip capace di analizzare rapidamente i batteri gram-positivi resistenti a tutti gli antibiotici.
«Il nostro obiettivo iniziale era di sviluppare uno strumento capace di identificare i geni di resistenza del batterio Bacillus anthracis, responsabile della malattia del carbonchio, una malattia primaria tra gli animali e pure utilizzata come arma biologica a scopi terroristici», precisa il professore Joachim Frey, codirettore del progetto.
«Ma il nostro incarico era pure di elaborare un programma più generale sulla sorveglianza delle resistenze agli antibiotici dei batteri presenti negli animali e potenzialmente trasmissibili all’essere umano».
Più rapido, più completo e meno caro
Il microchip è una novità mondiale, che apre nuove prospettive per i malati.
«Quando gli antibiotici non hanno più effetti sulle persone gravemente malate, la loro vita è rapidamente in pericolo. Alcuni ceppi batterici hanno infatti sviluppato una resistenza agli antibiotici e sono quindi diventati insensibili a certi farmaci, generando delle infezioni difficili da curare», indica il Fondo nazionale svizzero per la ricerca scientifica (FNS) in un comunicato.
Grazie a questa nuova tecnica, possono essere individuati i circa 90 geni di resistenza conosciuti per l’insieme dei batteri gram-positivi. «Sono questi che attualmente causano i problemi maggiori negli ospedali», prosegue il professore bernese.
Tra di essi vi sono gli agenti responsabili delle infezioni polmonari e cutanee, nonché setticemiche (stafilococchi, streptococchi) e tetaniche (clostridi).
Rispetto al metodo fenotipico utilizzato finora, questo nuovo test possiede un triplo vantaggio: è più rapido, più completo e costa meno.
«Questa nuova tecnica ci permette di determinare in qualche ora le resistenze più importanti», osserva Joachim Frey. «Ciò ci eviterà di curare degli uomini o degli animali con antibiotici contro i quali i germi sono resistenti». Finora, i geni dovevano essere ricercati uno dopo l’altro, un lavoro fastidioso che prendeva diversi giorni.
«Il microchip permette pure di scoprire dei geni ancora inattivi che il metodo fenotipico non può riconoscere».
Presto anche per i batteri gram-negativi
Potendo essere prodotto su larga scala, la «pulce» interessa in particolar modo il settore ospedaliero, ma anche l’industria animale e alimentare.
«Al centro della problematica vi è la sorveglianza delle epidemie, la comprensione di come questi geni di resistenza si trasferiscono da un corpo all’altro», spiega il professore dell’Università di Berna.
«Il test serve in effetti ad assicurare un’utilizzazione corretta degli antibiotici sugli animali da reddito, al fine di impedire la trasmissione del germe resistente alle derrate alimentari destinate alla consumazione umana», precisa da parte sua il FNS.
Ma esistono pure altre applicazioni importanti: «Questo nuovo strumento permette alle imprese del settore alimentare di analizzare i loro ceppi di batteri starters per i prodotti lattiferi, come lo yogurt o il formaggio», prosegue Joachim Frey.
Questa industria può così evitare che dei batteri portatori di germi di resistenza siano utilizzati nella produzione d’alimenti e che siano poi trasferiti all’essere umano.
Il microchip sarà prossimamente adattato al secondo grande gruppo: i batteri gram-negativi. «Dovrebbe vedere la luce il prossimo anno», afferma il ricercatore dell’Università di Berna.
Grazie a questo ulteriore sviluppo, potranno essere rapidamente individuati gli agenti responsabili delle infezioni al canale digestivo (salmonella, campilobatteri…), delle meningiti (meningococchi) e delle blenorragie (gonococchi).
Accesso diretto all’identità genetica
Professore assistente alla Facoltà di biologia e medicina dell’Università di Losanna e medico al Centro ospedaliero universitario vodese (CHUV), Giorgio Zanetti segue da vicino l’evoluzione di questi microchip.
«In futuro questo tipo di tecnologie si imporrà nella maggior parte dei laboratori – osserva –. Queste tecniche rappresentano un notevole risparmio di tempo. Oggi bisogna adattarsi al ciclo di vita delle colture prima di poterle analizzare. Con questi microchip, potremo interrogare direttamente l’identità genetica dei batteri».
Una scorciatoia che secondo il medico losannese è cruciale: «La disseminazione delle resistenza sarà rallentata. Questo aspetto è capitale, soprattutto in un ambiente ospedaliero dove bisogna evitare una trasmissione dei batteri».
L’epidemiologo emette tuttavia qualche riserva: «Questa rapidità ha un prezzo. Bisognerà perciò determinare in quali circostanze utilizzare queste nuove tecnologie affinché il tempo risparmiato giustifichi i costi supplementari».
swissinfo, Raphael Donzel
(traduzione di Daniele Mariani)
Batteri gram-positivi: tra di essi figurano gli agenti responsabili delle infezioni polmonari e cutanee, così come setticemiche (stafilococchi, streptococchi) e tetaniche (clostridi)
Batteri gram-negativi: Gli agenti responsabili delle infezioni del tubo digestivo (salmonella, campilobatteri…), delle meningiti (meningococchi) e delle blenorragie (gonococchi)
La ricerca dell’Università di Berna è stata effettuata in collaborazione con la società tedesca «Clondiag Chip Technologies GmbH» su mandato del Fondo nazionale svizzero.
La tecnologia «ADN microarray» utilizzata è identica a quella impiegata per numerosi test del DNA.
Per sviluppare il nuovo microchip, i ricercatori hanno repertoriato tutti i geni conosciuti di resistenza dei batteri gram-positivi.
Campioni di DNA corrispondenti a questi geni sono in seguito scelti e posati sul microchip.
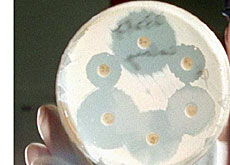
Il DNA del batterio da analizzare è isolato, marcato con un colorante e posato sul microchip. Se contiene un gene di resistenza identico a quelli precedentemente applicati sul microchip, resterà fissato sul microchip.
I punti colorati sono registrati da una telecamera e poi trasmessi al computer, che stabilisce il profilo di resistenza del ceppo batterico e alimenta una base dati.

In conformità con gli standard di JTI
Altri sviluppi: SWI swissinfo.ch certificato dalla Journalism Trust Initiative



Potete trovare una panoramica delle discussioni in corso con i nostri giornalisti Potete trovare una panoramica delle discussioni in corso con i nostri giornalisti qui.
Se volete iniziare una discussione su un argomento sollevato in questo articolo o volete segnalare errori fattuali, inviateci un'e-mail all'indirizzo italian@swissinfo.ch.